El test OncoLiq utiliza una tecnología que apunta a detectar en sangre la presencia de pequeñas moléculas llamadas «microARNs» liberadas por esos tumores apenas comienzan a producirse
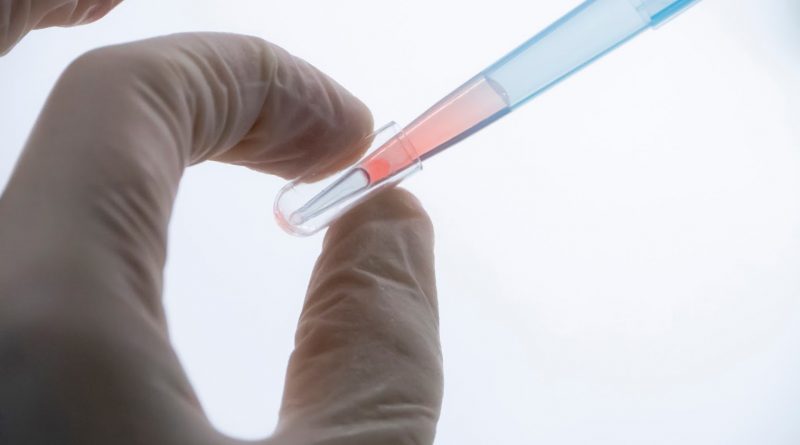
Un equipo de investigadores del Consejo Nacional de Investigaciones Científicas y Técnicas (Conicet) informó que avanza en el desarrollo del test Oncoliq, para la detección precoz del cáncer de mama, de mayor incidencia en mujeres, y de próstata, el de mayor incidencia en hombres, que apunta a “reducir las tasas de mortalidad de esos tumores mediante su identificación temprana con un análisis de sangre”.
“Oncoliq es de fácil implementación y permite detectar tempranamente el cáncer con alrededor de un 90% de sensibilidad, lo cual reduciría la mortalidad por cáncer y mejoraría la calidad de vida de los y las pacientes”, señaló una de las líderes del proyecto, investigadora del Conicet y directora del Laboratorio de Oncología Molecular y Nuevos Blancos Terapéuticos del Instituto de Biología y Medicina Experimental (Ibyme, Conicet-F-Ybyme), Adriana De Siervi.
El test utiliza una tecnología que apunta a detectar en sangre la presencia de pequeñas moléculas llamadas “microARNs” liberadas por esos tumores apenas comienzan a producirse, explicaron.
La validación de Oncoliq se realizó en 100 pacientes con cáncer y 100 voluntarios/as sanos/as, precisaron.
“Además hemos realizado una validación adicional que consistió en procesar con bioinformática datos de bases públicas de más de 2000 pacientes y voluntarios sanos con el fin de validar Oncoliq en poblaciones asiáticas confirmando la utilidad de este estudio a nivel global”, completó De Siervi.
Por otra parte, están realizando pruebas piloto en el Hospital Posadas, Hospital Militar Central, Hospital Melchor Posse, Sanatorio Otamendi, Cemafe, Instituto Alexander Fleming y Hospital Bernardo Houssay.
ONCOLiq
Hasta ahora se enrolaron 450 voluntarias para mama y 750 voluntarios para próstata, detallaron.
“El objetivo es llegar a mil reclutados/as para cada tipo de cáncer”, indicó De Siervi, quien además es doctora en Biología y lidera el proyecto junto a Marina Simian, investigadora del Conicet y directora del Laboratorio de Nanobiología del Instituto de Nanosistemas de la Universidad Nacional de San Martín (Unsam).
“Cuando los tumores de mama o de próstata son detectados tempranamente y están confinados dentro del órgano, el tratamiento es mucho menos agresivo en comparación con los que se detectan de manera tardía”, explicaron desde Conicet.
De Servi indicó que el objetivo es que Oncoliq “se aplique a personas que van a realizarse su control ginecológico o urológico anual”.
Y agregó que “a partir de esta muestra de sangre se utilizaría la técnica de PCR en tiempo real para detectar los microARNs y luego aplicando un algoritmo se informaría al individuo el resultado del ensayo. Es un test muy similar al test de Covid-19”.
Prueba piloto
Actualmente se está implementado una prueba piloto para determinar el valor predictivo positivo de Oncoliq en comparación con los estudios convencionales (mamografía, prueba del antígeno prostático específico o PSA según sus siglas y otros), detallaron.
“Es realmente emocionante saber que el trabajo de muchos becarios y estudiantes ha generado este test y actualmente exista esta Start up llamada Oncoliq que está generando, además de conocimiento de calidad, nuevas fuentes de trabajo para doctorados del mismo sistema científico”, concluyó la especialista.
El proyecto para desarrollar Oncoliq contó con subsidios de la Agencia Nacional de Promoción de la Investigación, el Desarrollo Tecnológico y la Innovación (Agencia I+D+i), el Instituto Nacional del Cáncer del Ministerio de Salud, la Fundación Williams y SF500, una iniciativa de Bioceres, la Provincia de Santa Fe y diversos actores que busca transformar investigadores e investigadoras en fundadores de startups en Ciencias de la Vida, concluyeron.
Fuente: Periferia
El Conicet desarrolla un test de detección precoz del cáncer de mama y de próstata












